2023年9月25日,浙江大学生物系统工程与食品科学学院何勇教授/冯旭萍副研究员团队与南京农业大学合作在Nature旗下植物学顶刊Nature Plants (IF5=18.6)在线发表了题为“Plantorganelle Hunter is an effective deep-learning-based method for plant organelle phenotyping in electron microscopy”的研究论文(Article),其他参与的研究院所还包括香港浸会大学、香港中文大学、墨尔本大学、名古屋大学等。这一突破性研究报道了全球首个在三维空间中重建植物亚细胞结构(细胞核、叶绿体、液泡、线粒体)的深度学习管道流程(Plantorganelle Hunter),创建的OrgSegNet模型首次实现了电镜数据上植物亚细胞结构精准分割,使植物亚细胞结构自动化三维重建成为现实。

细胞是生命活动的基本单位,其生命过程在复杂的三维空间中展开,各种细胞器在其中扮演着关键角色。然而,细胞器的形态和数量在不同生理状态下变化巨大,细胞器在细胞分裂、分化、衰老、外界胁迫等过程中发生一系列变化,以优化细胞器功能,因此,准确描述细胞器的三维形态并追踪其变化对于理解生命活动至关重要。
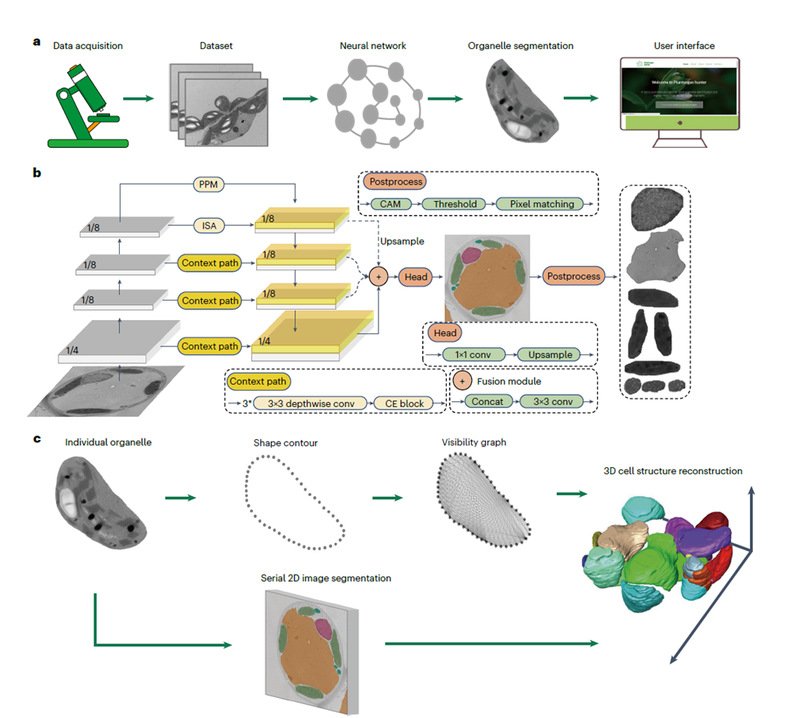
在这项研究中,研究团队首次开发了一种用于重建植物多种细胞器三维形态的深度学习管道流程Plantorganelle Hunter,可以对植物细胞器进行精确的三维重建和定量分析。为了提高模型的鲁棒性,团队采集了多达19种植物品种的透射电子显微镜图像,并标注了超过9000个细胞器用于OrgSegNet深度学习模型的构建。基于Plantorganelle Hunter和OrgSegNet能够实现多类植物细胞中包括叶绿体、细胞核、线粒体和液泡的精准分割,能够改变原有三维细胞器复杂且繁琐的重建过程,将这一过程从数十乃至数百小时缩短到几十分钟。在完整细胞中对细胞器的快速三维重建有望加速对空间中细胞相互作用的生物过程和组织的理解,这项技术的成功应用为植物学研究提供了新的思路和方法。目前,研究团队已经开源了所有的算法和数据,并且热情地邀请相关领域的研究者一起来探索和补充这些数据,相信这将加速计算科学和生物领域对细胞活动的深入理解和新发现。
该研究是农业工程、植物科学和计算机科学多学科交叉融合的研究成果。论文第一单位为浙江大学生物系统工程与食品科学学院,浙江大学DAAI团队冯旭萍副研究员和硕士研究生俞泽宇为论文共同一作,浙江大学生物系统工程和食品科学学院何勇教授和南京农业大学生命科学学院刘峰副教授为通讯作者。该研究项目得到了国家自然科学基金、浙江省重点研发计划等项目的资助。
原文链接:https://doi.org/10.1038/s41477-023-01527-5

